一, Taasan ang sensitivity ng sistema ng reaksyon:
1. Ihiwalay ang mataas na kalidad na RNA:
Ang matagumpay na synthesis ng cDNA ay nagmumula sa mataas na kalidad na RNA.Ang mataas na kalidad na RNA ay dapat na hindi bababa sa buong haba at walang reverse transcriptase inhibitors gaya ng EDTA o SDS.Tinutukoy ng kalidad ng RNA ang maximum na dami ng sequence information na maaari mong i-transcribe sa cDNA.Ang karaniwang paraan ng paglilinis ng RNA ay isang one-step na paraan gamit ang guanidine isothiocyanate/acid phenol.Upang maiwasan ang kontaminasyon sa pamamagitan ng mga bakas na halaga ng RNase, ang RNA na nakahiwalay sa mga sample na mayaman sa RNase (gaya ng pancreas) ay kailangang itago sa formaldehyde upang mapanatili ang mataas na kalidad na RNA, lalo na para sa pangmatagalang imbakan.Ang RNA na nakuha mula sa atay ng daga ay karaniwang nasira pagkatapos na maimbak sa tubig sa loob ng isang linggo, habang ang RNA na nakuha mula sa pali ng daga ay nananatiling matatag pagkatapos na maimbak sa tubig sa loob ng 3 taon.Bilang karagdagan, ang mga transcript na mas mahaba sa 4 kb ay mas sensitibo sa pagkasira ng mga trace RNases kaysa sa maliliit na transcript.Upang mapataas ang katatagan ng mga nakaimbak na sample ng RNA, ang RNA ay maaaring matunaw sa deionized formamide at maiimbak sa -70°C.Ang formamide na ginamit upang mapanatili ang RNA ay dapat na walang RNA-degrading debris.Ang RNA mula sa pancreas ay maaaring mapanatili sa formamide nang hindi bababa sa isang taon.Kapag naghahanda na gumamit ng RNA, maaari mong gamitin ang sumusunod na paraan upang mamuo ang RNA: idagdag ang NaCl sa 0.2M at 4 na beses ang dami ng ethanol, ilagay sa temperatura ng silid sa loob ng 3-5 minuto, at centrifuge sa 10,000×g sa loob ng 5 minuto.
2. Gamitin ang RNaseH-inactive (RNaseH-) reverse transcriptase:
Ang mga inhibitor ng RNase ay kadalasang idinaragdag sa reverse transcription reactions upang mapataas ang haba at ani ng cDNA synthesis.Ang mga inhibitor ng RNase ay dapat idagdag sa panahon ng reaksyon ng synthesis ng unang strand sa pagkakaroon ng isang buffer at isang ahente ng pagbabawas (tulad ng DTT), dahil ang proseso bago ang synthesis ng cDNA ay nagde-denature sa inhibitor, sa gayon ay naglalabas ng nakagapos na RNase na maaaring magpababa ng RNA.Pinipigilan lamang ng mga inhibitor ng protina RNase ang pagkasira ng RNA ng RNase A, B, C, at hindi pinipigilan ang RNase sa balat, kaya mag-ingat na huwag ipasok ang RNase mula sa iyong mga daliri sa kabila ng paggamit ng mga inhibitor na ito.
Ang reverse transcriptase ay nag-catalyze ng conversion ng RNA sa cDNA.Ang parehong M-MLV at AMV ay may endogenous na aktibidad ng RNaseH bilang karagdagan sa kanilang sariling aktibidad ng polymerase.Ang aktibidad ng RNaseH at aktibidad ng polymerase ay nakikipagkumpitensya sa isa't isa para sa hybrid strand na nabuo sa pagitan ng RNA template at ng DNA primer o cDNA extension strand, at pinapababa ang RNA strand sa RNA:DNA complex.Ang template ng RNA na pinababa ng aktibidad ng RNaseH ay hindi na maaaring magsilbi bilang isang epektibong substrate para sa synthesis ng cDNA, na binabawasan ang ani at haba ng cDNA synthesis.Samakatuwid, magiging kapaki-pakinabang na alisin o lubos na bawasan ang aktibidad ng RNaseH ng reverse transcriptase.
Ang SuperScript Ⅱ reverse transcriptase, RNaseH- MMLV reverse transcriptase at thermoScript reverse transcriptase, RNaseH- AMV, ay maaaring makakuha ng mas maraming halaga at mas full-length na cDNA kaysa sa MMLV at AMV.Ang sensitivity ng RT-PCR ay maaapektuhan ng dami ng cDNA synthesis.Ang ThermoScript ay mas sensitibo kaysa sa AMV.Ang laki ng mga produktong RT-PCR ay limitado sa pamamagitan ng kakayahan ng reverse transcriptase na mag-synthesize ng cDNA, lalo na kapag nag-clone ng mas malalaking cDNA.Kung ikukumpara sa MMLV, ang SuperScripⅡ ay tumaas nang malaki sa ani ng mahabang produkto ng RT-PCR.Ang RNaseH-reverse transcriptase ay mayroon ding tumaas na thermostability, kaya ang reaksyon ay maaaring gawin sa mga temperatura na mas mataas kaysa sa normal na 37-42°C.Sa ilalim ng mga iminungkahing kondisyon ng synthesis, gumamit ng oligo(dT) primer at 10 μCi ng [α-P]dCTP.Ang kabuuang ani ng unang strand ay kinakalkula gamit ang TCA precipitation method.Ang buong-haba na cDNA ay nasuri gamit ang laki-sorted bands excised at binibilang sa isang alkaline agarose gel.
3. Taasan ang incubation temperature para sa reverse transcription:
Ang isang mas mataas na temperatura ng pagpapapisa ng itlog ay nakakatulong upang buksan ang pangalawang istraktura ng RNA, na nagdaragdag ng ani ng reaksyon.Para sa karamihan ng mga template ng RNA, ang pag-incubate sa RNA at mga primer sa 65°C na walang buffer o asin, na sinusundan ng mabilis na paglamig sa yelo ay aalisin ang karamihan sa mga pangalawang istruktura at hahayaan ang mga primer na magbigkis.Gayunpaman, ang ilang mga template ay mayroon pa ring pangalawang istruktura, kahit na pagkatapos ng denaturation ng init.Maaaring gawin ang amplification ng mahihirap na template na ito gamit ang ThermoScript Reverse Transcriptase at paglalagay ng reverse transcription reaction sa mas mataas na temperatura upang mapabuti ang amplification.Ang mas mataas na temperatura ng incubation ay maaari ding tumaas ang pagiging tiyak, lalo na kapag ang mga gene-specific primers (GSP) ay ginagamit para sa cDNA synthesis (tingnan ang Kabanata 3).Kung gumagamit ng GSP, tiyaking ang Tm ng mga panimulang aklat ay pareho sa inaasahang temperatura ng incubation.Huwag gumamit ng oligo(dT) at mga random na panimulang aklat sa itaas ng 60°C.Ang mga random na primer ay nangangailangan ng incubation sa 25°C sa loob ng 10 minuto bago tumaas sa 60°C.Bilang karagdagan sa paggamit ng mas mataas na reverse transcription temperature, maaari ding mapabuti ang specificity sa pamamagitan ng direktang paglilipat ng RNA/primer mix mula sa 65°C denaturation temperature patungo sa reverse transcription incubation temperature at pagdaragdag ng pre-warmed 2× reaction mixture (cDNA hot-start synthesis) .Nakakatulong ang diskarteng ito na maiwasan ang intermolecular base pairing na nangyayari sa mas mababang temperatura.Ang maraming pagbabago sa temperatura na kinakailangan para sa RT-PCR ay maaaring gawing simple sa pamamagitan ng paggamit ng thermal cycler.
Ang Tth thermostable polymerase ay gumaganap bilang isang DNA polymerase sa presensya ng Mg2+ at bilang isang RNA polymerase sa presensya ng Mn2+.Maaari itong panatilihing mainit-init sa pinakamataas na temperatura na 65°C.Gayunpaman, ang pagkakaroon ng Mn2+ sa panahon ng PCR ay binabawasan ang katapatan, na ginagawang hindi angkop ang Tth polymerase para sa high-precision na amplification, tulad ng pag-clone ng cDNA.Bilang karagdagan, ang Tth ay may mababang reverse transcription na kahusayan, na nagpapababa ng sensitivity, at, dahil ang reverse transcription at PCR ay maaaring gawin gamit ang isang enzyme, ang mga control reaction na walang reverse transcription ay hindi maaaring gamitin upang ihambing ang mga produkto ng amplification ng cDNA na may kontaminadong genomic DNA.Ang mga produkto ng amplification ay pinaghiwalay.
4. Mga additives na nagsusulong ng reverse transcription:
Ang mga additives kabilang ang glycerol at DMSO ay idinagdag sa first-strand synthesis reaction, na maaaring mabawasan ang katatagan ng nucleic acid double-strand at makalas ang pangalawang istraktura ng RNA.Hanggang 20% glycerol o 10% DMSO ang maaaring idagdag nang hindi naaapektuhan ang aktibidad ng SuperScript II o MMLV.Maaari ding tiisin ng AMV ang hanggang 20% glycerol nang walang pagkawala ng aktibidad.Upang ma-maximize ang sensitivity ng RT-PCR sa SuperScriptⅡ reverse transcription reaction, 10% glycerol ay maaaring idagdag at incubated sa 45°C.Kung ang 1/10 ng reverse transcription reaction product ay idinagdag sa PCR, kung gayon ang konsentrasyon ng gliserol sa amplification reaction ay 0.4%, na hindi sapat upang pigilan ang PCR.
5. RNaseH treat:
Ang paggamot sa mga reaksyon ng synthesis ng cDNA na may RNaseH bago ang PCR ay maaaring magpapataas ng sensitivity.Para sa ilang mga template, iniisip na ang RNA sa cDNA synthesis reaction ay pumipigil sa pagbubuklod ng mga produkto ng amplification, kung saan ang paggamot sa RNaseH ay maaaring magpapataas ng sensitivity.Sa pangkalahatan, kinakailangan ang paggamot sa RNaseH kapag pinapalaki ang mas mahabang full-length na mga template ng target ng cDNA, gaya ng low-copy tuberous scherosis II.Para sa mahirap na template na ito, pinahusay ng paggamot sa RNaseH ang signal na ginawa ng SuperScript II o AMV-synthesized cDNA.Para sa karamihan ng mga reaksyon ng RT-PCR, opsyonal ang paggamot sa RNaseH, dahil ang hakbang ng denaturation ng PCR sa 95°C ay karaniwang nag-hydrolyze sa RNA sa RNA:DNA complex.
6. Pagpapabuti ng Paraan ng Maliit na RNA Detection:
Ang RT-PCR ay lalong mapaghamong kapag maliit na halaga ng RNA ang magagamit.Ang glycogen na idinagdag bilang isang carrier sa panahon ng RNA isolation ay nakakatulong upang mapataas ang ani ng maliliit na sample.Ang RNase-free glycogen ay maaaring idagdag kasabay ng pagdaragdag ng Trizol.Ang glycogen ay nalulusaw sa tubig at maaaring panatilihin sa may tubig na bahagi na may RNA upang tulungan ang kasunod na pag-ulan.Para sa mga sample na mas mababa sa 50 mg ng tissue o 106 cultured cell, ang inirerekomendang konsentrasyon ng RNase-free glycogen ay 250 μg/ml.
Ang pagdaragdag ng acetylated BSA sa reverse transcription reaction gamit ang SuperScript II ay maaaring tumaas ang sensitivity, at para sa maliit na halaga ng RNA, ang pagbabawas sa dami ng SuperScript II at pagdaragdag ng 40 unit ng RNaseOut nuclease inhibitor ay maaaring tumaas ang antas ng pagtuklas.Kung ang glycogen ay ginagamit sa proseso ng paghihiwalay ng RNA, inirerekomenda pa rin na magdagdag ng BSA o RNase inhibitor kapag gumagamit ng SuperScript II para sa reverse transcription reaction.
二、Taasan ang pagtitiyak ng RT-PCR
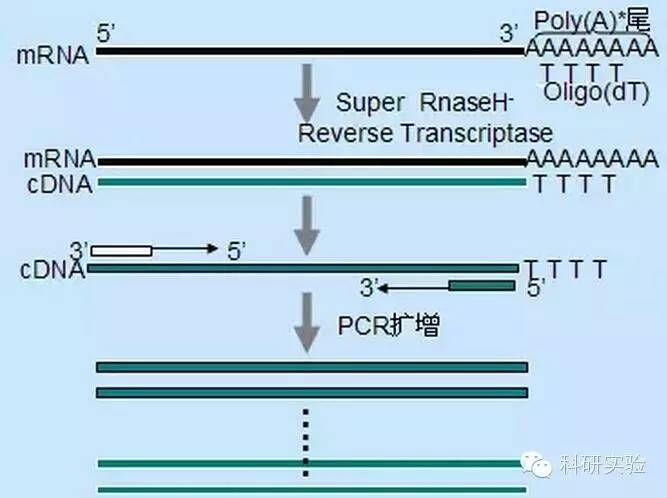
1. CND Asynthesis:
Maaaring simulan ang first-strand cDNA synthesis gamit ang tatlong magkakaibang pamamaraan, ang relatibong pagtitiyak nito ay nakakaapekto sa dami at uri ng cDNA na na-synthesize.
Ang random na paraan ng panimulang aklat ay ang hindi gaanong tiyak sa tatlong pamamaraan.Ang mga panimulang aklat ay sumisira sa maraming mga site sa buong transcript, na bumubuo ng maikli, bahagyang-haba na mga cDNA.Ang pamamaraang ito ay madalas na ginagamit upang makakuha ng 5′ na mga pagkakasunud-sunod ng pagtatapos at upang makakuha ng cDNA mula sa mga template ng RNA na may mga rehiyon ng pangalawang istraktura o may mga site ng pagwawakas na hindi maaaring kopyahin ng reverse transcriptase.Upang makuha ang pinakamahabang cDNA, ang ratio ng mga primer sa RNA sa bawat sample ng RNA ay kailangang matukoy nang empirically.Ang panimulang konsentrasyon ng mga random na primer ay mula 50 hanggang 250 ng bawat 20 μl na reaksyon.Dahil ang cDNA na na-synthesize mula sa kabuuang RNA gamit ang mga random na primer ay pangunahing ribosomal RNA, ang poly(A)+RNA ay karaniwang pinipili bilang template.
Ang mga primer na Oligo(dT) ay mas tiyak kaysa sa mga random na primer.Nag-hybrid ito sa poly(A) tail na matatagpuan sa 3′ dulo ng karamihan sa mga eukaryotic mRNA.Dahil ang poly(A)+ RNA ay humigit-kumulang 1% hanggang 2% ng kabuuang RNA, ang halaga at pagiging kumplikado ng cDNA ay mas mababa kaysa sa mga random na primer.Dahil sa mataas na pagtitiyak nito, ang oligo(dT) sa pangkalahatan ay hindi nangangailangan ng pag-optimize ng ratio ng RNA sa mga primer at poly(A)+ na seleksyon.Inirerekomenda na gumamit ng 0.5μg oligo(dT) bawat 20μl reaction system.Ang oligo(dT)12-18 ay angkop para sa karamihan ng RT-PCR.Ang ThermoScript RT-PCR System ay nag-aalok ng oligo(dT)20 dahil sa mas magandang thermal stability nito para sa mas mataas na temperatura ng incubation.
Ang Gene specific primers (GSP) ay ang pinakaspesipikong primer para sa reverse transcription step.Ang GSP ay isang antisense oligonucleotide na maaaring partikular na mag-hybrid sa RNA target sequence, hindi tulad ng mga random na primer o oligo(dT), na sumasama sa lahat ng RNA.Ang parehong mga patakaran na ginamit upang magdisenyo ng mga PCR primer ay nalalapat sa disenyo ng GSP sa mga reverse transcription reaction.Ang GSP ay maaaring maging kapareho ng pagkakasunud-sunod ng amplification primer na sumasama sa 3′-pinaka dulo ng mRNA, o ang GSP ay maaaring idisenyo upang i-anneal sa ibaba ng agos ng reverse amplification primer.Para sa ilang amplified na paksa, higit sa isang antisense primer ang kailangang idisenyo para sa matagumpay na RT-PCR dahil ang pangalawang istraktura ng target na RNA ay maaaring pumigil sa primer binding.Inirerekomenda na gumamit ng 1 pmol antisense GSP sa isang 20 μl first-strand synthesis reaction.
2. Taasan ang incubation temperature para sa reverse transcription:
Upang lubos na mapakinabangan ang buong bentahe ng pagtitiyak ng GSP, dapat gumamit ng reverse transcriptase na may mas mataas na thermostability.Ang mga thermotable na reverse transcriptases ay maaaring i-incubate sa mas mataas na temperatura upang mapataas ang higpit ng reaksyon.Halimbawa, kung ang isang GSP ay na-anne sa 55°C, ang pagiging tiyak ng GSP ay hindi ganap na magagamit kung ang AMV o M-MLV ay ginagamit para sa reverse transcription sa mababang stringency na 37°C.Gayunpaman, ang SuperScript II at ThermoScript ay maaaring mag-react sa 50°C o mas mataas, na mag-aalis ng mga hindi partikular na produkto na nabuo sa mas mababang temperatura.Para sa maximum na specificity, ang RNA/primer mix ay maaaring ilipat nang direkta mula sa 65°C denaturation temperature patungo sa reverse transcription incubation temperature at idagdag sa isang pre-warmed 2× reaction mix (cDNA synthesis hot start).Nakakatulong ito na maiwasan ang pagpapares ng intermolecular base sa mababang temperatura.Ang maraming pagbabago sa temperatura na kinakailangan para sa RT-PCR ay maaaring gawing simple sa pamamagitan ng paggamit ng thermal cycler.
3. Binabawasan ang genomic DNA contamination:
Ang isang potensyal na kahirapan na nakatagpo sa RT-PCR ay ang kontaminasyon ng genomic DNA sa RNA.Ang paggamit ng isang mahusay na paraan ng paghihiwalay ng RNA, tulad ng Trizol Reagent, ay magbabawas sa dami ng genomic DNA na nakakahawa sa paghahanda ng RNA.Upang maiwasan ang mga produktong nagmula sa genomic DNA, ang RNA ay maaaring tratuhin ng amplification-grade DNase I upang maalis ang kontaminadong DNA bago ang reverse transcription.Ang DNase I digestion ay tinapos sa pamamagitan ng pagpapapisa ng mga sample sa 2.0 mM EDTA sa loob ng 10 minuto sa 65 ° C.Maaaring i-chelate ng EDTA ang mga magnesium ions, na pumipigil sa magnesium ion-dependent RNA hydrolysis sa mataas na temperatura.
Upang paghiwalayin ang amplified na cDNA mula sa pagkontamina ng genomic DNA amplification na mga produkto, ang mga primer ay maaaring idisenyo na ang bawat isa ay mag-anne sa paghihiwalay ng mga exon.Ang mga produktong PCR na nagmula sa cDNA ay magiging mas maikli kaysa sa mga nakuha mula sa kontaminadong genomic DNA.Bilang karagdagan, ang isang eksperimento sa kontrol na walang reverse transcription ay isinagawa sa bawat template ng RNA upang matukoy kung ang isang ibinigay na fragment ay nagmula sa genomic DNA o cDNA.Ang produktong PCR na nakuha nang walang reverse transcription ay nagmula sa genome.
Oras ng post: Mayo-16-2023








