Kasama sa eksperimento sa RT-qPCR ang pagkuha ng RNA at pagtatasa ng kalidad, reverse transcription at qPCR tatlong hakbang, bawat hakbang ay may maraming pag-iingat, ipakikilala namin nang detalyado sa ibaba.
Ⅰ.Pagtatasa ng kalidad ng RNA
Sa eksperimento ng RT-qPCR, pagkatapos makumpleto ang pagkuha ng RNA, kailangang suriin ang kalidad ng RNA, at ang follow-up na eksperimento ay maaari lamang isagawa pagkatapos na maging kwalipikado.Kasama sa mga pamamaraan ng pagsusuri ang spectrophotometer, Agilent gel electrophoresis, Agilent 2100 analysis, kung saan ang pinakakaraniwang ginagamit na spectrophotometer at agarose gel electrophoresis method detection.Dapat tandaan na ang dalawang pamamaraan na ito ay kailangang gamitin nang magkasama upang makumpleto ang pagtuklas at pagsusuri ng konsentrasyon, kadalisayan at integridad ng RNA, upang matiyak ang kalidad ng RNA.
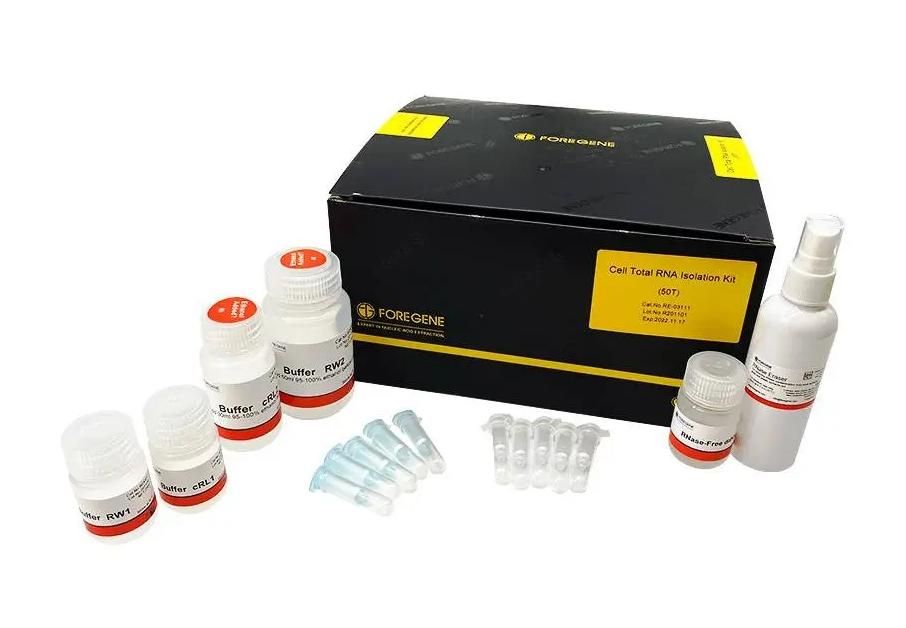
Kaugnay na RNA Isolation Kit:
Ang mataas na purified at mataas na kalidad na kabuuang RNA ay maaaring makuha mula sa iba't ibang kulturang mga cell sa loob ng 11min.
Animal Total RNA Isolation Kit
Mabilis at mahusay na i-extract ang high-purity at de-kalidad na kabuuang RNA mula sa iba't ibang tissue ng hayop.
Spectrophotometer:
Ang spectrophotometer ay pangunahing ginagamit upang matukoy ang konsentrasyon at kadalisayan ng RNA, ngunit hindi nito makita ang integridad ng RNA at genomic residue.Kabilang sa mga ito, ang A260/280 at A260/230 ay mahalagang mga parameter para sa pagtuklas ng kadalisayan ng RNA, at ang kadalisayan ng RNA ay maaaring makita ayon sa pagbabagu-bago ng kanilang mga halaga:
1. 1.9< A260/280< 2.1, na nagpapahiwatig na ang kadalisayan ng RNA ay mabuti;A260/280<1.9, na nagpapahiwatig na maaaring may nalalabi sa protina sa RNA;A260/280>2.1, na nagpapahiwatig ng posibleng bahagyang pagkasira ng RNA, na maaaring higit pang makumpirma ng agarose gel electrophoresis.
2. 2.0< A260/230< 2.2, na nagpapahiwatig na ang kadalisayan ng RNA ay mabuti;A260/230< 2.0, na nagpapahiwatig na maaaring may mga nalalabi ng mga organic na reagents sa RNA, tulad ng mga phenol, ethanol o sugars.
Agarose gel electrophoresis:
Maaaring pag-aralan ng agarose gel electrophoresis assay ang integridad ng RNA, mga residue ng genome at protina, ngunit hindi tumpak na mabibilang ang konsentrasyon ng RNA o matukoy ang mga nalalabi ng mga organikong reagents.Kunin ang mga template ng eukaryotic RNA bilang halimbawa:
1. Ang RNA ay sumailalim sa agarose gel electrophoresis.Kung mayroon lamang tatlong solong banda ng 28sRNA, 18sRNA at 5.8sRNA sa mapa ng gel, ito ay nagpapahiwatig na ang nakuhang RNA ay buo.Kung mayroong isang pag-drag na kababalaghan, ito ay nagpapahiwatig ng bahagyang pagkasira ng RNA.
2. Kung mayroong isang maliwanag na banda sa pagitan ng butas ng pandikit at ng 28sRNA band, maaaring mayroong genomic DNA residue.
3. Kung lumilitaw ang mga banda sa butas ng kola, ito ay nagpapahiwatig na maaaring may mga nalalabi ng protina at iba pang mga macromolecular na sangkap.
Ⅱ. Baliktad na transkripsyon
Matapos makumpleto ang pagkuha ng RNA, kailangan itong i-reverse sa cDNA para sa mga kasunod na eksperimento, kaya mahalaga ang hakbang sa pagbabalik.Ang reverse transcription ay ipakikilala mula sa pagpili ng reverse transcriptase at primer:
Baliktarin ang pagpili ng transcriptase:
Kasama sa mga karaniwang reverse transcriptases ang AMV RTase at MMLV RTase.Ang RNase H ng AMV RTase ay may malakas na aktibidad, maikling haba ng synthesis, mababang halaga ng synthesis at mahusay na thermal stability (42 ~ 55 ℃).Ang aktibidad ng RNase H ng MMLV RTase ay mahina, ang haba ng synthesis ay mahaba, ang halaga ng synthesis ay mataas, at ang thermal stability ay mahina (37 ~ 42 ℃).
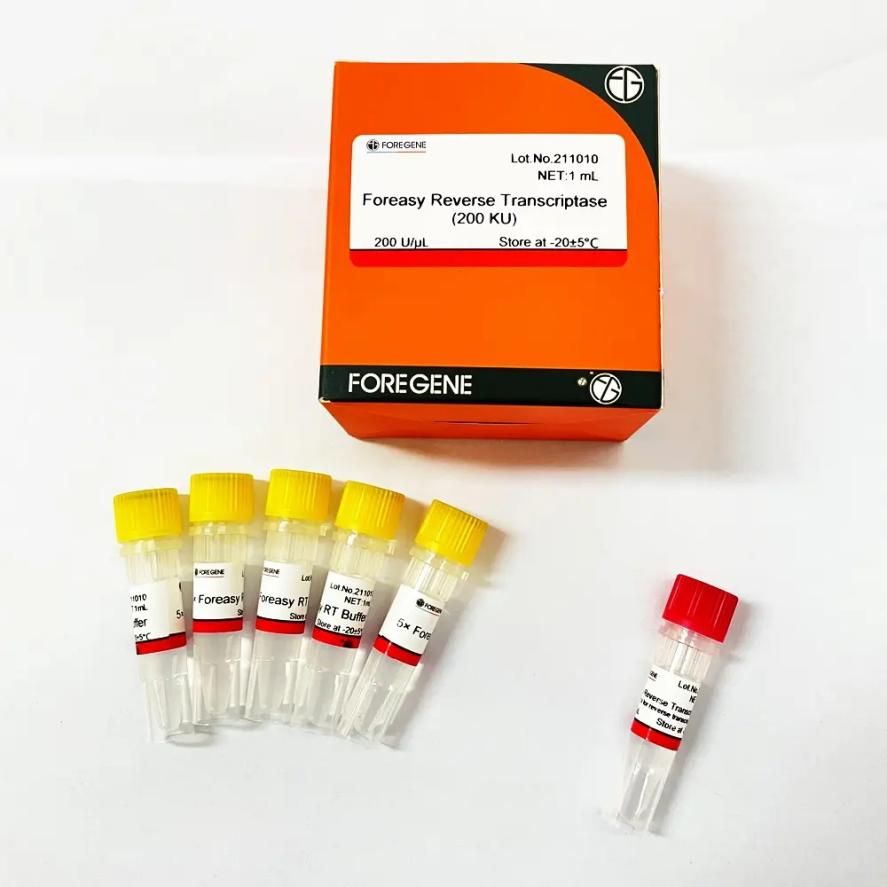
Dahil ang RNase H enzyme ay may function ng degrading RNA template, ang MMLV na may mahinang RNase H na aktibidad ay dapat na mas pinili sa panahon ng reverse transcription, at pagkatapos ng genetic engineering, ang thermal stability ng MMLV ay umabot sa isang qualitative leap.Pagkuha ng ForegeneForeasy Reverse Transcriptase(M-MLV para sa reverse transcription) bilang halimbawa, ito ay isang bagong reverse transcriptase na ipinahayag sa E. coli engineered bacteria gamit ang genetic recombination technology.Ito ay isang recombinant DNA polymerase na nag-synthesize ng isang komplementaryong DNA strand mula sa single-stranded RNA, DNA, o isang RNA:DNA hybrid.Wala itong aktibidad ng RNase H, malakas na katatagan, malakas na RNA affinity, at mataas na sensitivity ng pagtuklas.
Foreasy Reverse Transcriptase(M-MLV para sa reverse transcription)
Pagpili ng panimulang aklat:
Sa pangkalahatan, nahahati ang mga primer ng RT sa tatlong kategorya: oligo dT, mga random na primer, at mga primer na partikular sa gene.Pumili ng angkop na mga panimulang aklat para sa paggamit ayon sa iba't ibang mga pang-eksperimentong kinakailangan.
1. Kung ang template ay eukaryotic na pinagmulan at ang huling cDNA ay ginagamit para sa regular na PCR amplification, Oligo (dT) ay inirerekomenda;Kung ang kasunod na eksperimento ay ginagamit lamang para sa qPCR, ang Oligo (dT) ay inirerekomenda na ihalo sa mga random na primer upang mapabuti ang kahusayan ng reverse transcription.
2. Kung ang template ay mula sa mga prokaryote, ang Random Primer o gene specific primer ay dapat piliin para sa reverse transcription.
Ⅲ.qPCR
Ang pagsukat ng fluorescence ay higit sa lahat ay pinapaliwanag mula sa pagpili ng mga pamamaraan ng dami, mga prinsipyo ng disenyo ng panimulang aklat, pagpili ng ROX, pagsasaayos ng system ng reaksyon at setting ng mga kondisyon ng reaksyon, atbp.
Pagpili ng mga pamamaraan ng dami:
Ang mga pamamaraan ng dami ay nahahati sa mga kamag-anak na pamamaraan ng dami at mga pamamaraan ng ganap na dami.Maaaring gamitin ang relative quantification upang makita ang epekto ng ilang mga pamamaraan ng paggamot sa expression ng gene, makita ang pagkakaiba ng expression ng gene sa iba't ibang oras at ihambing ang pagkakaiba ng expression ng gene sa iba't ibang mga tissue.Maaaring matukoy ng absolute quantification ang dami ng nucleic acid sa virus at iba pa.Kapag gumagawa ng mga eksperimento, dapat nating piliin ang naaangkop na mga pamamaraan ng dami ayon sa sarili nating mga eksperimento.
Mga prinsipyo ng pangunahing disenyo:
Ang disenyo ng panimulang aklat para sa qPCR ay direktang nauugnay sa kahusayan ng amplification at pagtitiyak ng produkto.Samakatuwid, ang wastong pagdidisenyo ng magagandang primer ay ang unang hakbang ng matagumpay na qPCR.Sa disenyo ng panimulang aklat, ang mga sumusunod na prinsipyo ay dapat bigyang-pansin kapag natutugunan ang prinsipyo ng maginoo na disenyo ng panimulang aklat:
1. Ang haba ng target na fragment ay kinokontrol sa pagitan ng 100 at 300 bp;
2. Cross-exon na disenyo upang maiwasan ang impluwensya ng genomic DNA;
3. Ang mga dinisenyong panimulang aklat ay kailangang masuri para sa kahusayan ng amplification, at kapag ang kahusayan ng amplification ay umabot sa pamantayan (90-110%) maaari silang magamit para sa dami ng mga eksperimento;
4. Ang konsentrasyon ng panimulang aklat ay karaniwang na-optimize sa pagitan ng 0.1uM at 1.0uM.
Pagpipilian ngROX:
Sa proseso ng quantitative reaction, ang ROX ay maaaring ayusin ang optical path difference, pipetting error o volume difference na dulot ng evaporation at condensation nang pantay, pagpapabuti ng repeatability ng mga resulta.Gayunpaman, dapat tandaan na ang pagpili ng ROX ay nauugnay sa instrumento.Kung ang instrumento ng qPCR ay may function na awtomatikong itama ang pagkakaiba sa pagitan ng mga butas, hindi nito kailangang magdagdag ng ROX;kung hindi, kailangan nitong magdagdag ng ROX correction.Ang mga maliliit na kasosyo sa pagbili ng mga reagents ay dapat na ayon sa instrumento na ginamit upang piliin ang tamang ROX, maiwasan ang mga pagkakamali sa ibang pagkakataon.
Paghahanda ng sistema ng reaksyon:
Mas gusto ang dami ng reaksyon na 20ul at 50ul.Ang mga sumusunod na bagay ay dapat bigyang pansin kapag nabuo ang sistema:
1. Ang sistema ng reaksyon ay kailangang ihanda sa pamamagitan ng bentilasyon sa ultra-clean na workbench, bagong ddH2Ginagamit ang O para sa bawat eksperimento;
2. Kailangang ihanda ng bawat eksperimento ang NTC para i-verify kung may polusyon sa system, at bawat pares ng mga panimulang aklat ay kailangang gumawa ng NTC kapag inihahanda ang system;
3. Upang matukoy kung mayroong gDNA residue sa RNA template, maaaring ihanda ang NRT para sa bawat sample para sa pagtuklas;
4. Kapag inihahanda ang sistema, inirerekumenda na gumawa ng hindi bababa sa 3 teknikal na pag-uulit para sa isang sample;
5. Kapag ang template ay cDNA, inirerekumenda na maghalo ng 5-10 beses upang mabawasan ang epekto ng pagsugpo ng reverse transcription system sa qPCR experiment.Mas mainam na galugarin ang dami ng template sa pamamagitan ng gradient, upang ang halaga ng CT ay bumaba sa pagitan ng 20-30;
6. Tukuyin ang kinakailangang bilang ng mga reaksyon, dagdagan ng 5-10% batay sa bilang ng mga reaksyon, at kalkulahin ang numero ng pagsasaayos ng volume;
7, ang sistema ay inihanda gamit ang prinsipyo ng premix, paghahalo pagkatapos ng sentripugasyon at matiyak na walang mga bula;
8, Bilang malayo hangga't maaari upang pumili ng sumusuporta sa mga consumable.
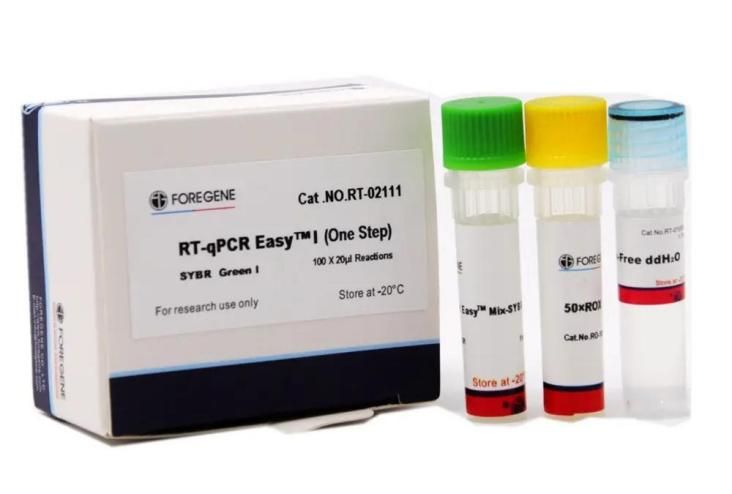
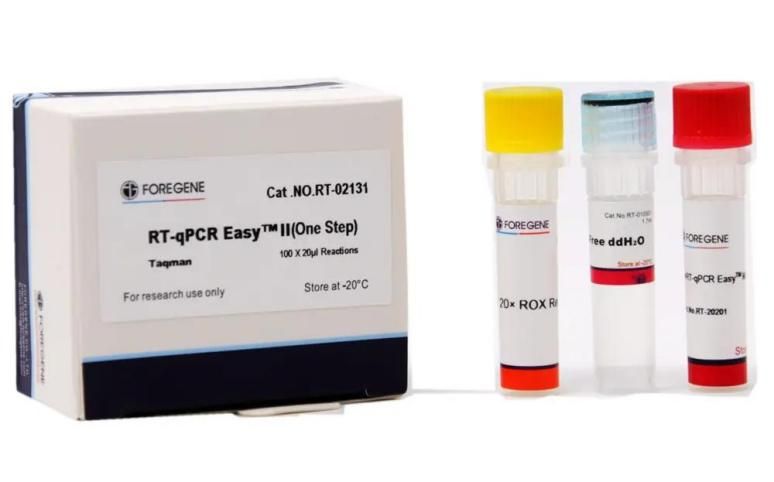
Kaugnay na RT-qPCR Kit
Gumagamit ang kit ng isang natatanging Foregene reverse transcription reagent at Foregene HotStar Taq DNA Polymerase na pinagsama sa isang natatanging sistema ng reaksyon upang epektibong mapabuti ang kahusayan sa amplification at pagtitiyak ng reaksyon.
Oras ng post: Abr-23-2023